library("sf")
library("terra")
library("dplyr")
library("readr")
library("ggplot2")
library("PerformanceAnalytics")
library("pastecs")
library("car")
library("psych")BiEc4_N Multivariate Modelle
Einstieg Multivariate Modelle / Habitatselektionsmodell
Libraries laden
Aufgabe 1
Einlesen des Gesamtdatensatzes für die Multivariate Analyse von Moodle
- Sichtung des Datensatzes, der Variablen und der Datentypen
- Kontrolle wieviele Rehe in diesem Datensatz enthalten sind
Code
DF_mod <- read_delim("datasets/fallstudie_n/Aufgabe4_Datensatz_Habitatnutzung_Modelle_231027_moodle.csv", delim = ";")
str(DF_mod)
class(DF_mod$time_of_day)
table(DF_mod$id)
DF_mod |>
group_by(id) |>
summarize(anzahl = n())
length(unique(DF_mod$id))Aufgabe 2
Unterteilung des Datensatzes in Teildatensätze entsprechend der Tageszeit
Code
DF_mod_night <- DF_mod |>
filter(time_of_day == "night")
DF_mod_day <- DF_mod |>
filter(time_of_day == "day")
# Kontrolle
table(DF_mod_night$time_of_day)
##
## night
## 3964
table(DF_mod_day$time_of_day)
##
## day
## 3964Aufgabe 3
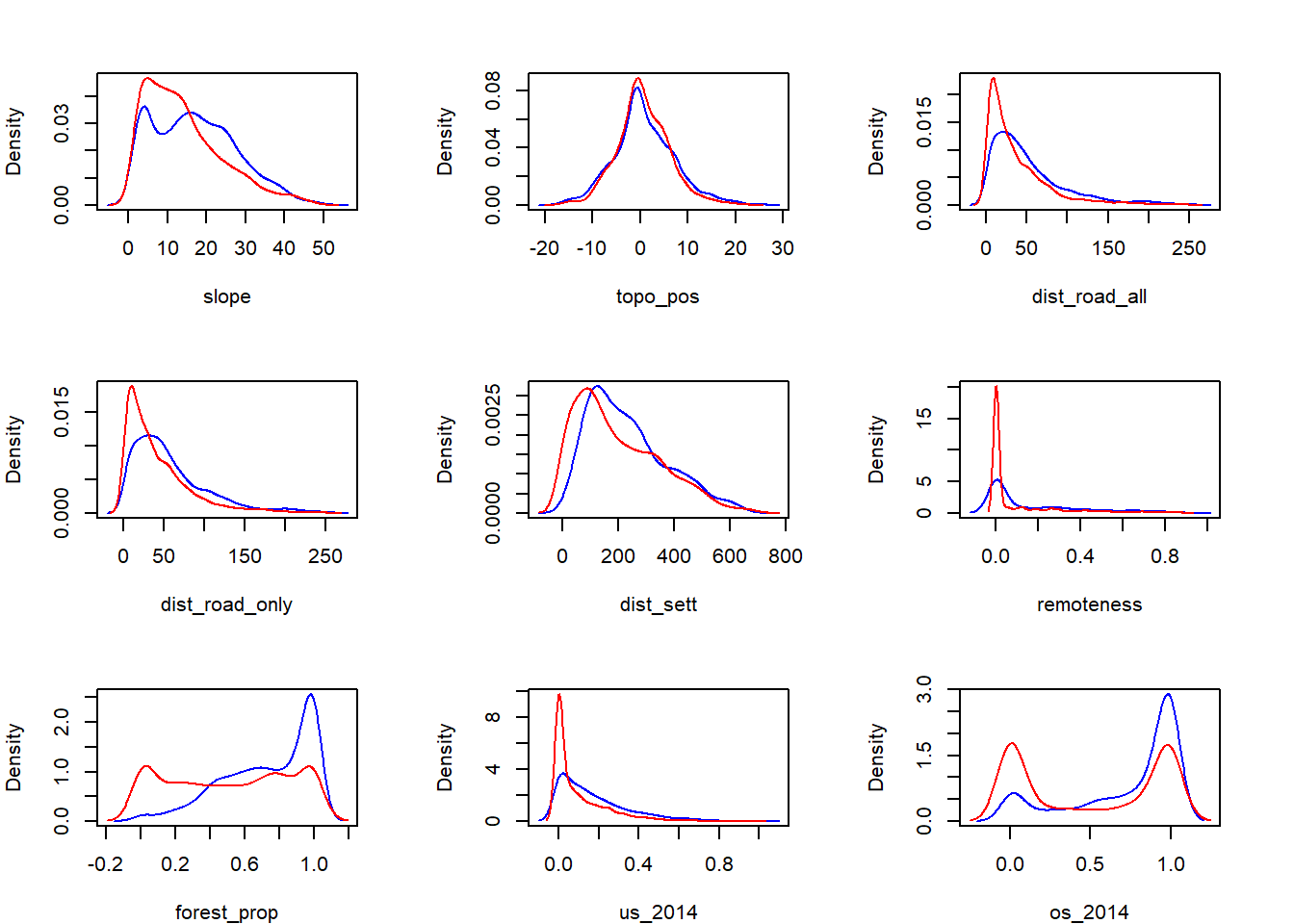
Erstellen von Density Plots der Präsenz / Absenz in Abhängigkeit der unabhängigen Variablen. Diese Übung dient einer ersten groben Einschätzung der Wirkung der Umweltvariablen auf die abhängige Variable (Präsenz/Absenz in unserem Fall)
# Ein Satz Density Plots für den Tagesdatensatz und einer für den Nachtdatensatz
par(mfrow = c(3, 3), mar = c(4, 4, 3, 3)) # Vorbereitung Raster für Plots
# innerhalb des for()-loops die Nummern der gewünschten Spalten einstellen
for (i in 6:14) {
dp <- DF_mod_day |> filter(pres_abs == 1) |> pull(i)
dp <- density(dp)
da <- DF_mod_day |> filter(pres_abs == 0) |> pull(i)
da <- density(da)
plot(0, 0, type = "l",
xlim = range(c(dp$x, da$x)),
ylim = range(dp$y, da$y),
xlab = names(DF_mod_day[i]),
ylab = "Density"
)
lines(dp$x, dp$y, col = "blue") # Präsenz
lines(da$x, da$y, col = "red") # Absenz
}
Aufgabe 4
Testen eurer erklärenden Variablen auf Normalverteilung (nur kontinuierliche)
Code
# klassischer Weg mit shapiro-wilk (mehrere Spalten, verschiedenene statistische
# Kenngrössen werden angezeigt. Normalverteilung: Wert ganz unten. p>0.05 = ja)
stat.desc(DF_mod_day[6:14], basic = F, norm = T)
## slope topo_pos dist_road_all dist_road_only
## median 1.370633e+01 2.327148e-01 2.986606e+01 3.505609e+01
## mean 1.535619e+01 7.331813e-01 4.261936e+01 4.765685e+01
## SE.mean 1.629186e-01 9.339098e-02 6.676922e-01 6.901438e-01
## CI.mean.0.95 3.194122e-01 1.830989e-01 1.309052e+00 1.353070e+00
## var 1.052144e+02 3.457351e+01 1.767202e+03 1.888047e+03
## std.dev 1.025741e+01 5.879925e+00 4.203811e+01 4.345166e+01
## coef.var 6.679657e-01 8.019742e+00 9.863616e-01 9.117611e-01
## skewness 7.330597e-01 2.782704e-01 1.872269e+00 1.631521e+00
## skew.2SE 9.424641e+00 3.577606e+00 2.407098e+01 2.097578e+01
## kurtosis -9.231943e-02 7.476704e-01 4.033217e+00 2.994463e+00
## kurt.2SE -5.936055e-01 4.807452e+00 2.593322e+01 1.925412e+01
## normtest.W 9.419360e-01 9.902728e-01 8.067698e-01 8.431235e-01
## normtest.p 6.839535e-37 7.269676e-16 2.675211e-56 1.245661e-52
## dist_sett remoteness forest_prop us_2014
## median 1.809658e+02 0.000000e+00 6.554673e-01 5.882353e-02
## mean 2.119112e+02 1.188578e-01 5.976772e-01 1.194621e-01
## SE.mean 2.358616e+00 3.228686e-03 5.272256e-03 2.386145e-03
## CI.mean.0.95 4.624214e+00 6.330041e-03 1.033659e-02 4.678188e-03
## var 2.205200e+04 4.132237e-02 1.101861e-01 2.256978e-02
## std.dev 1.484992e+02 2.032790e-01 3.319428e-01 1.502324e-01
## coef.var 7.007614e-01 1.710270e+00 5.553882e-01 1.257574e+00
## skewness 7.209630e-01 1.808899e+00 -4.046525e-01 1.647457e+00
## skew.2SE 9.269118e+00 2.325625e+01 -5.202447e+00 2.118066e+01
## kurtosis -1.740088e-01 2.374388e+00 -1.115659e+00 2.923840e+00
## kurt.2SE -1.118861e+00 1.526710e+01 -7.173587e+00 1.880002e+01
## normtest.W 9.452976e-01 6.538165e-01 9.104534e-01 7.934120e-01
## normtest.p 4.868489e-36 3.078496e-67 2.141161e-43 1.674851e-57
## os_2014
## median 7.847363e-01
## mean 5.939976e-01
## SE.mean 6.653214e-03
## CI.mean.0.95 1.304404e-02
## var 1.754675e-01
## std.dev 4.188884e-01
## coef.var 7.052022e-01
## skewness -4.252236e-01
## skew.2SE -5.466921e+00
## kurtosis -1.571720e+00
## kurt.2SE -1.010602e+01
## normtest.W 7.807897e-01
## normtest.p 1.397443e-58
# empfohlener Weg
ggplot(DF_mod_day, aes(slope)) +
geom_histogram(aes(y = after_stat(density)), color = "black", fill = "white") +
stat_function(fun = dnorm, args = list(mean = mean(DF_mod_day$slope, na.rm = T), sd = sd(DF_mod_day$slope, na.rm = T)), color = "black", size = 1)
Code
# Aufgabe 4: die Verteilung bei einem Teildatensatz zu testen reicht,
# denn die verwendeten Kreise sind die selben am Tag und in der Nacht,
# nur die Nutzung durch das Reh nichtAufgabe 5
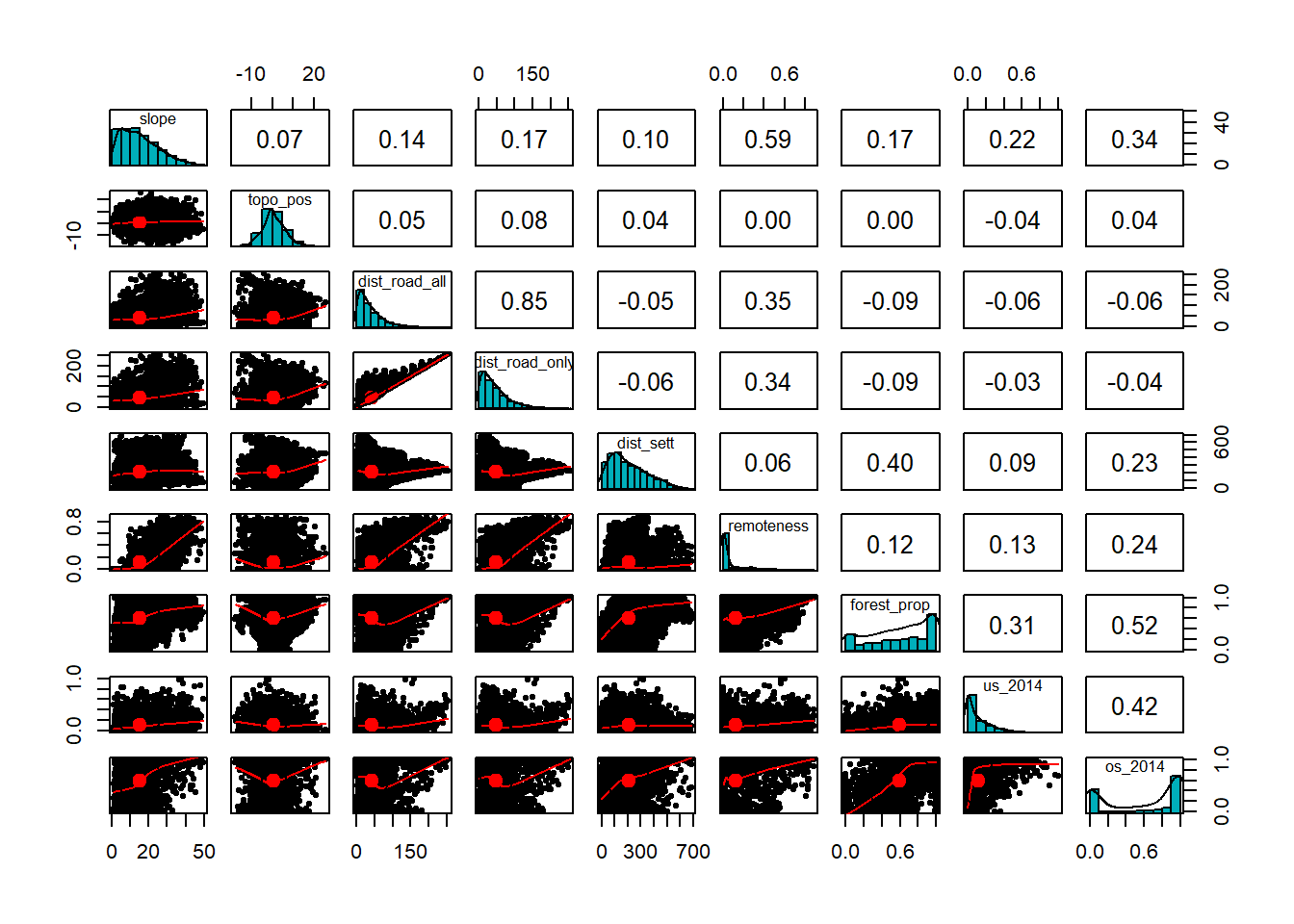
Explorative Analysen der Variablen mit Scatterplots / Scatterplotmatrizen
- Zu Scatterplots und Scatterplotmatrizen gibt es viele verschiedene Funktionen / Packages, schaut im Internet und sucht euch eines welches euch passt.
- Testen der Korrelation zwischen den Variablen (Parametrisch oder nicht-parametrische Methode? Ausserdem: gewisse Scatterplotmatrizen zeigen euch die Koeffizenten direkt an)
Code
chart.Correlation(DF_mod_day[6:14], histogram = TRUE, pch = 19, method = "kendall")
Code
# ?chart.Correlation
pairs.panels(DF_mod_day[6:14],
method = "kendall", # correlation method
hist.col = "#00AFBB",
density = TRUE, # show density plots
ellipses = TRUE # show correlation ellipses
)
Code
# Aufgabe 5: die Korrelation bei einem Teildatensatz zu testen reicht,
# denn die verwendeten Kreise sind die selben am Tag und in der Nacht,
# nur die Nutzung durch das Reh nicht.