library("sf")
library("terra")
library("dplyr")
library("readr")
library("ggplot2")
library("PerformanceAnalytics")
library("pastecs")
library("car")
library("psych")
library("DHARMa")
options(scipen = 999)Multivariate Modelle
Einstieg Multivariate Modelle / Habitatselektionsmodell
Libraries laden
Aufgabe 1
Einlesen der Gesamtdatensätze für die Multivariate Analysen von Moodle
- Sichtung der Datensätze, der Variablen und der Datentypen
- Kontrolle wieviele Rehe/Rothirsche in den Datensätzen enthalten sind
Musterlösung
DF_reh <- read_delim("datasets/fallstudie_n/Aufgabe_4/Aufgabe4_Datensatz_Modelle_Habitatnutzung_Reh_251027.csv", delim = ";")
str(DF_reh)
class(DF_reh$time_of_day)
table(DF_reh$id)
DF_reh |>
group_by(id) |>
summarize(anzahl = n())
length(unique(DF_reh$id))Aufgabe 2
Unterteilung des Datensatzes in Teildatensätze entsprechend der Tageszeit
Musterlösung
DF_reh_night <- DF_reh |>
filter(time_of_day == "night")
DF_reh_day <- DF_reh |>
filter(time_of_day == "day")Aufgabe 3
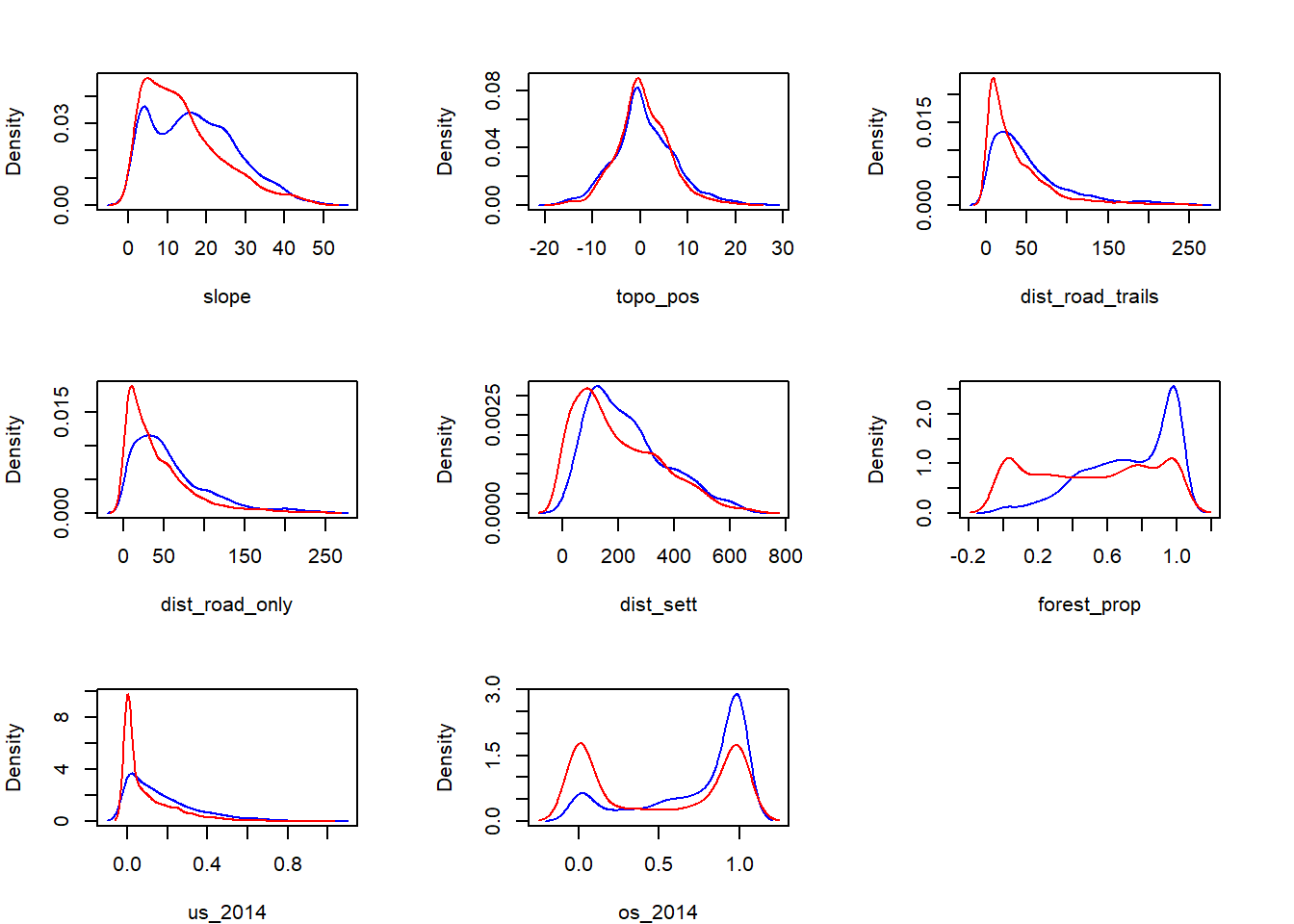
Erstellen von Density Plots der Präsenz / Absenz in Abhängigkeit der unabhängigen Variablen. Diese Übung dient einer ersten groben Einschätzung der Wirkung der Umweltvariablen auf die abhängige Variable (Präsenz/Absenz in unserem Fall)
# Ein Satz Density Plots für den Tagesdatensatz und einer für den Nachtdatensatz
par(mfrow = c(3, 3), mar = c(4, 4, 3, 3)) # Vorbereitung Raster für Plots
# innerhalb des for()-loops die Nummern der gewünschten Spalten einstellen
for (i in 5:12) {
dp <- DF_reh_day |> filter(pres_abs == 1) |> pull(i)
dp <- density(dp)
da <- DF_reh_day |> filter(pres_abs == 0) |> pull(i)
da <- density(da)
plot(0, 0, type = "l",
xlim = range(c(dp$x, da$x)),
ylim = range(dp$y, da$y),
xlab = names(DF_reh_day[i]),
ylab = "Density"
)
lines(dp$x, dp$y, col = "blue") # Präsenz = used
lines(da$x, da$y, col = "red") # Absenz = available
}
Aufgabe 4
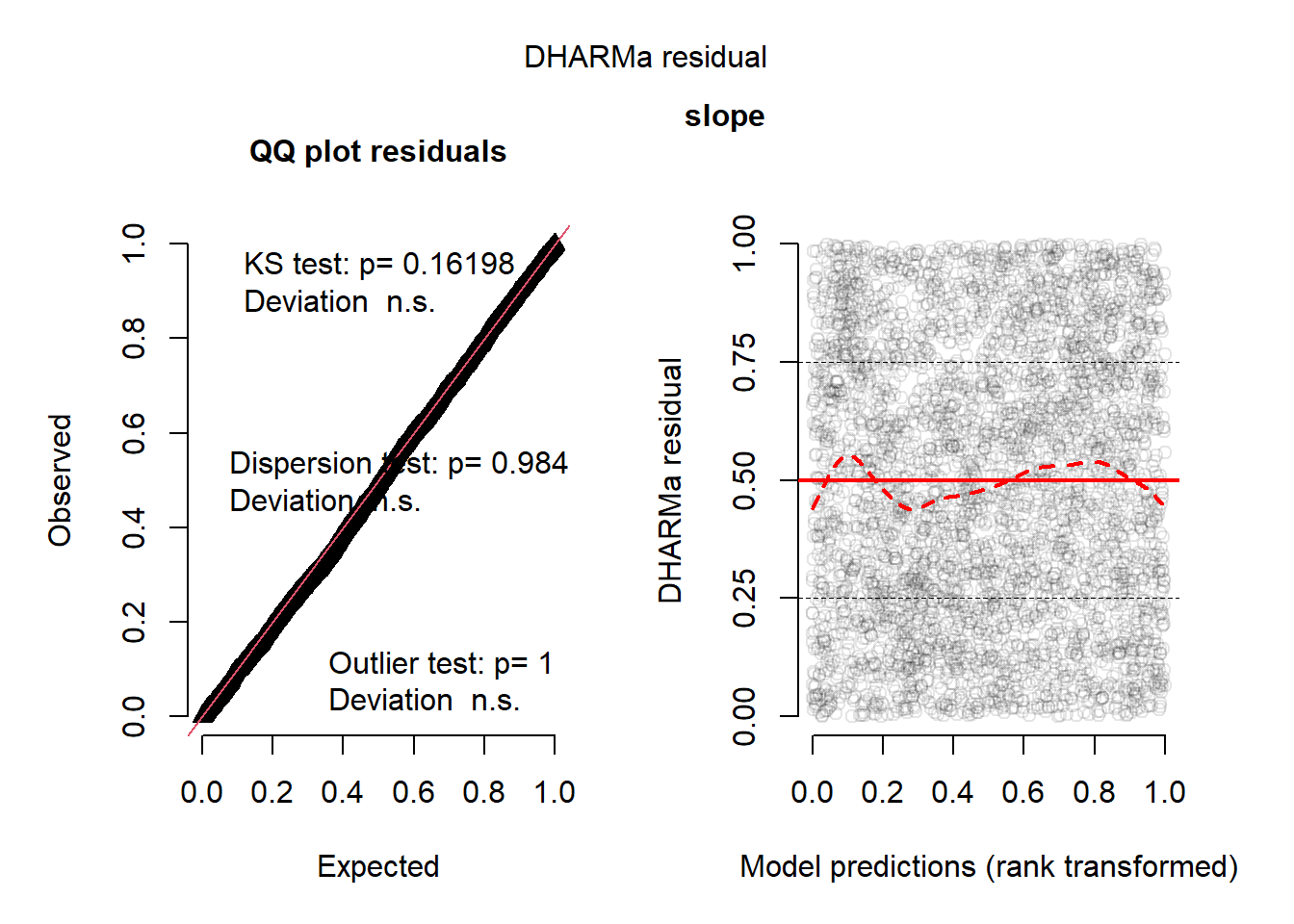
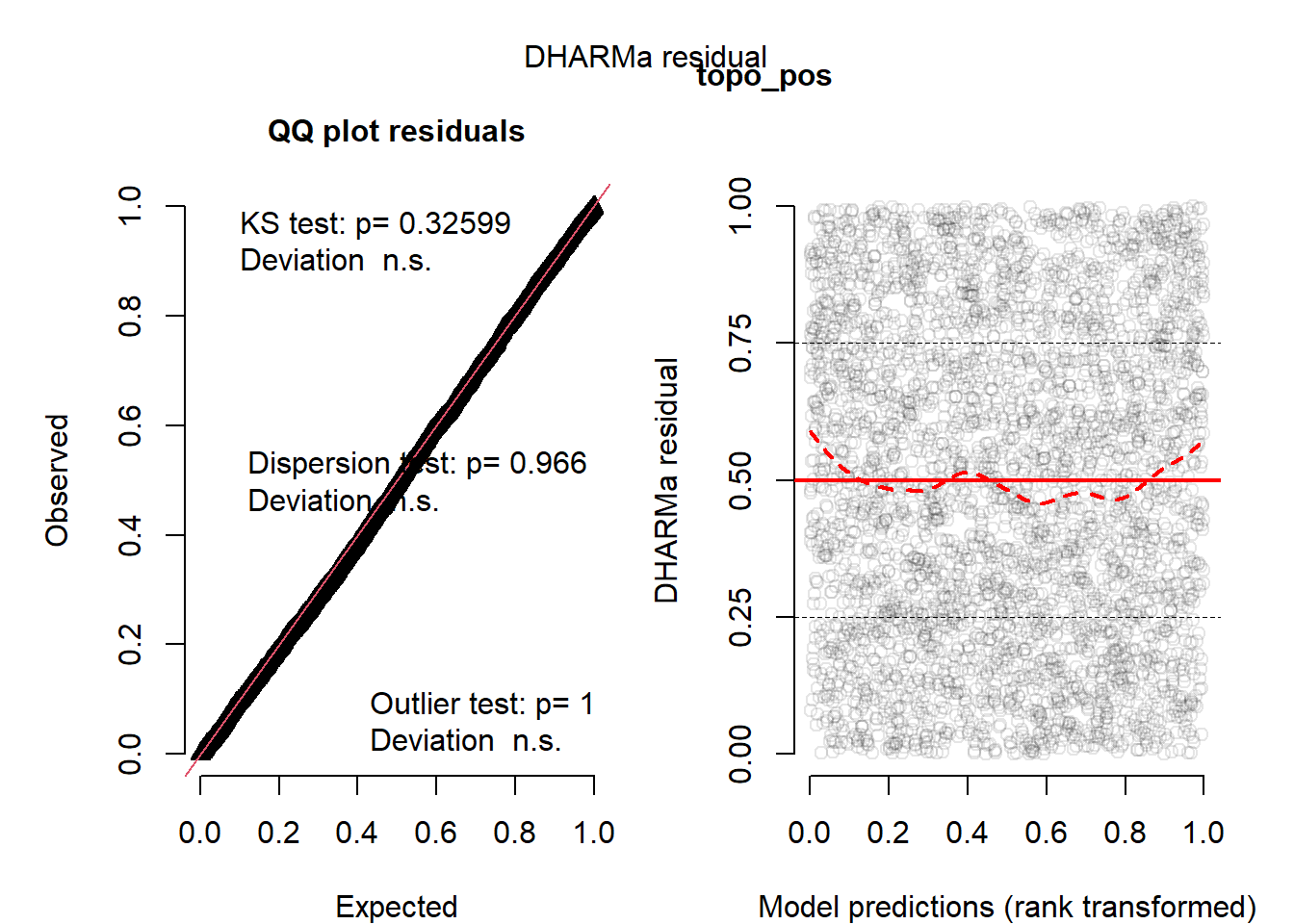
Für jede kontinuierliche Variable: ein binomiales GLM fitten (pres_abs ~ x, data = DF_mod_day, family = binomial), Residuen berechnen und ein Histogramm + Q-Q-Plot (Residuen sollen grob „normal“ wirken) plotten. Wenn starke Abweichungen auftreten (krummes Histogramm, Q-Q-Punkte weit von der Linie): Transformationen probieren, das Modell neu fitten und die Residuen nochmal überprüfen.
Musterlösung
# Definition des zu verarbeitenden Teildatensatzes
data <- DF_reh_day
# Liste mit den zu verarbeitenden kontinuierlichen Variablen
vars <- c("slope","topo_pos","dist_road_trails","dist_road_only",
"dist_sett","forest_prop","us_2014","os_2014")
# Ausgabe-Datei als PDF
out_pdf <- "fallstudie_n/glm_DHARMa_checks_DF_reh_day.pdf"
pdf(out_pdf, width = 7, height = 6)
on.exit(dev.off(), add = TRUE)
# Loop über die Variablen
for (v in vars) {
if (!v %in% names(data)) next
x <- data[[v]]
if (sum(!is.na(x)) < 30 || length(unique(na.omit(x))) < 3) next
# GLM fitten
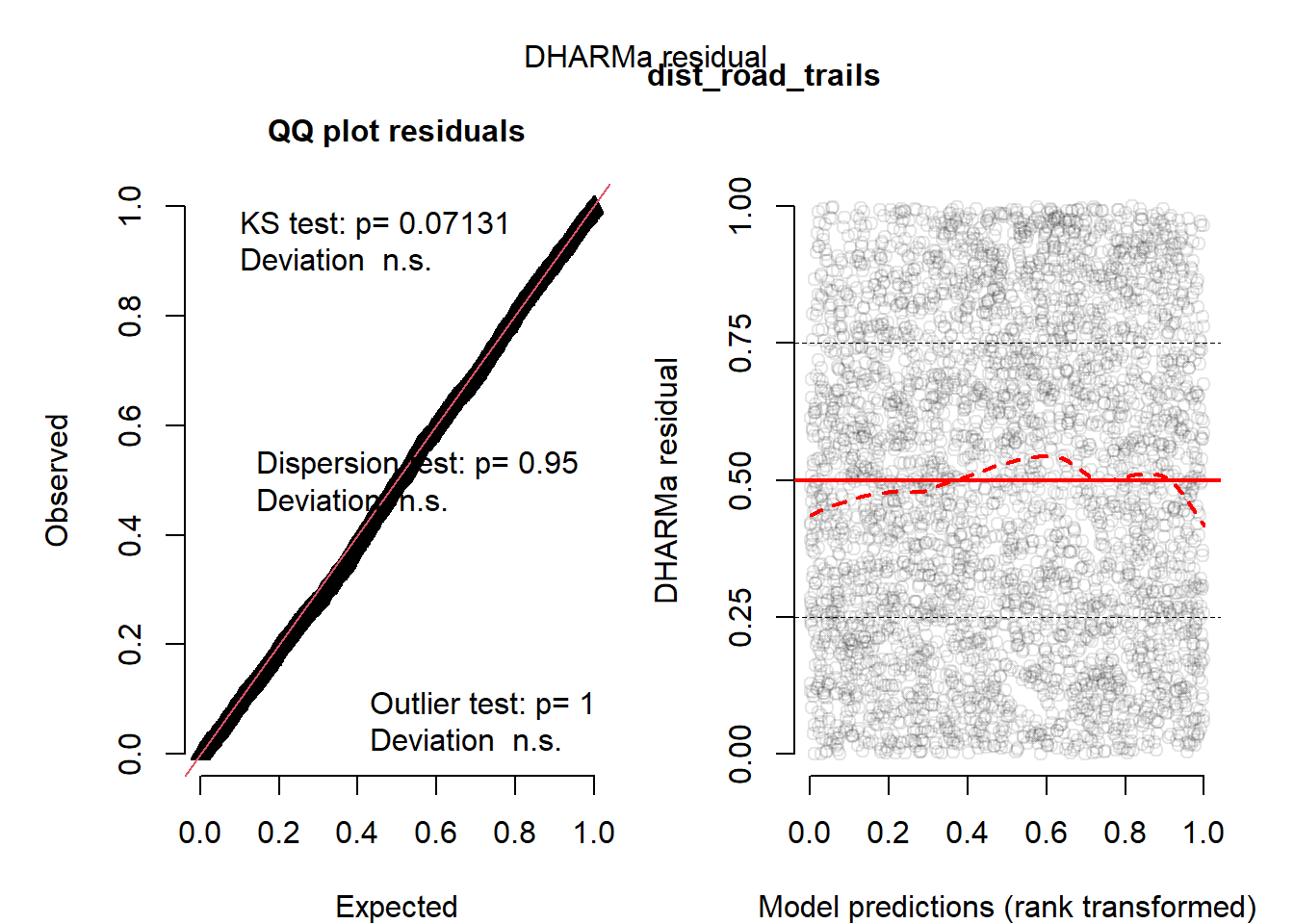
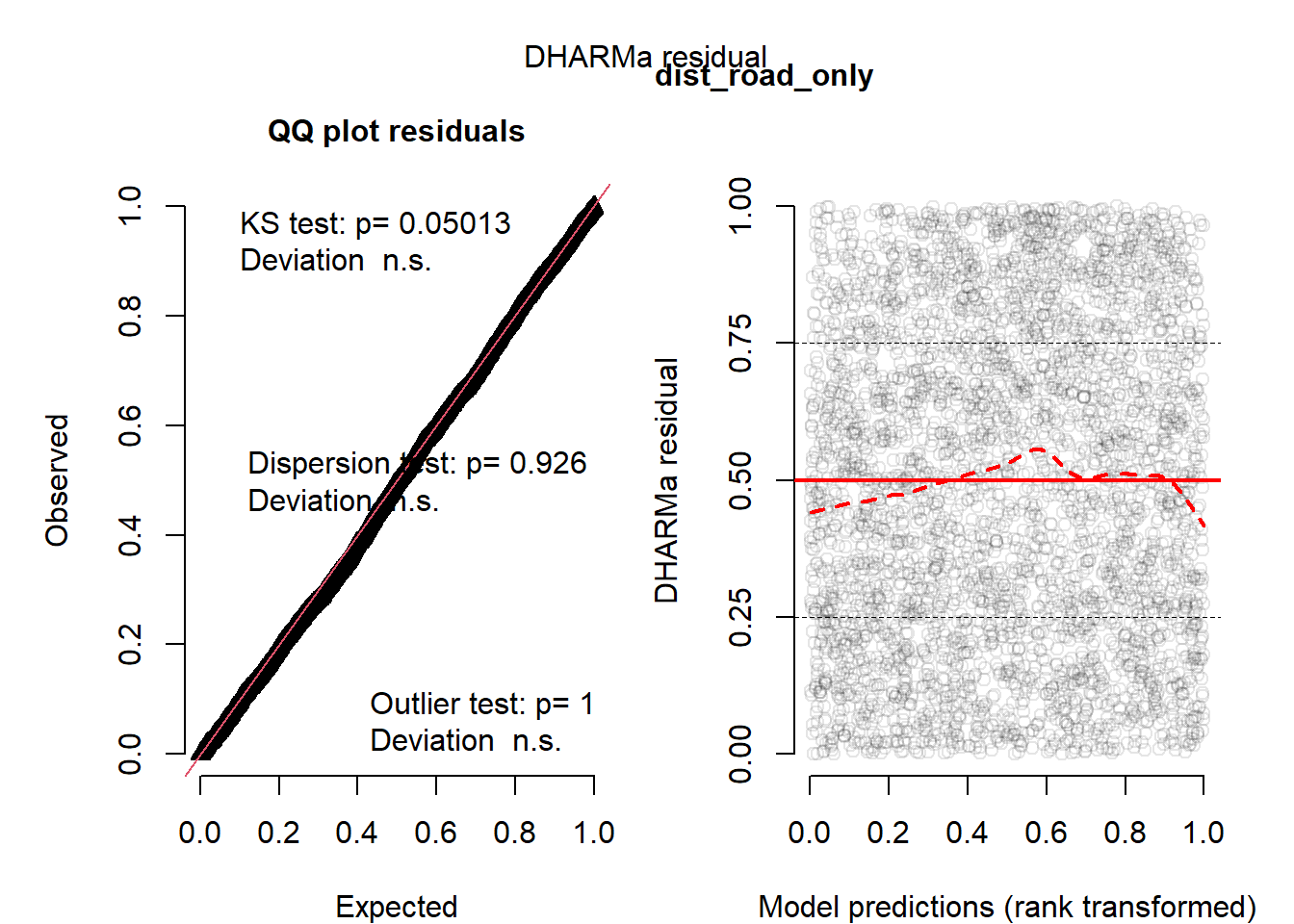
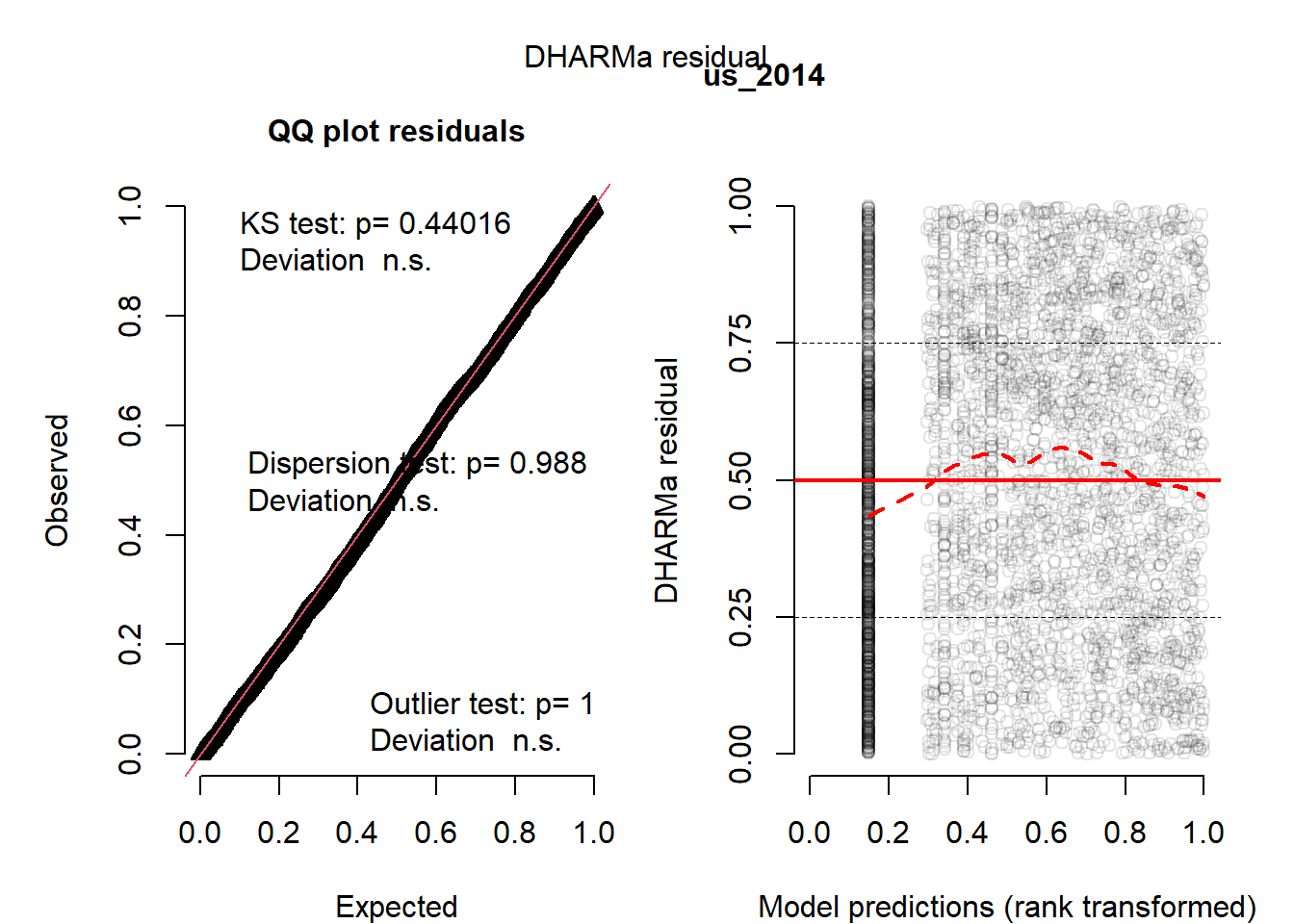
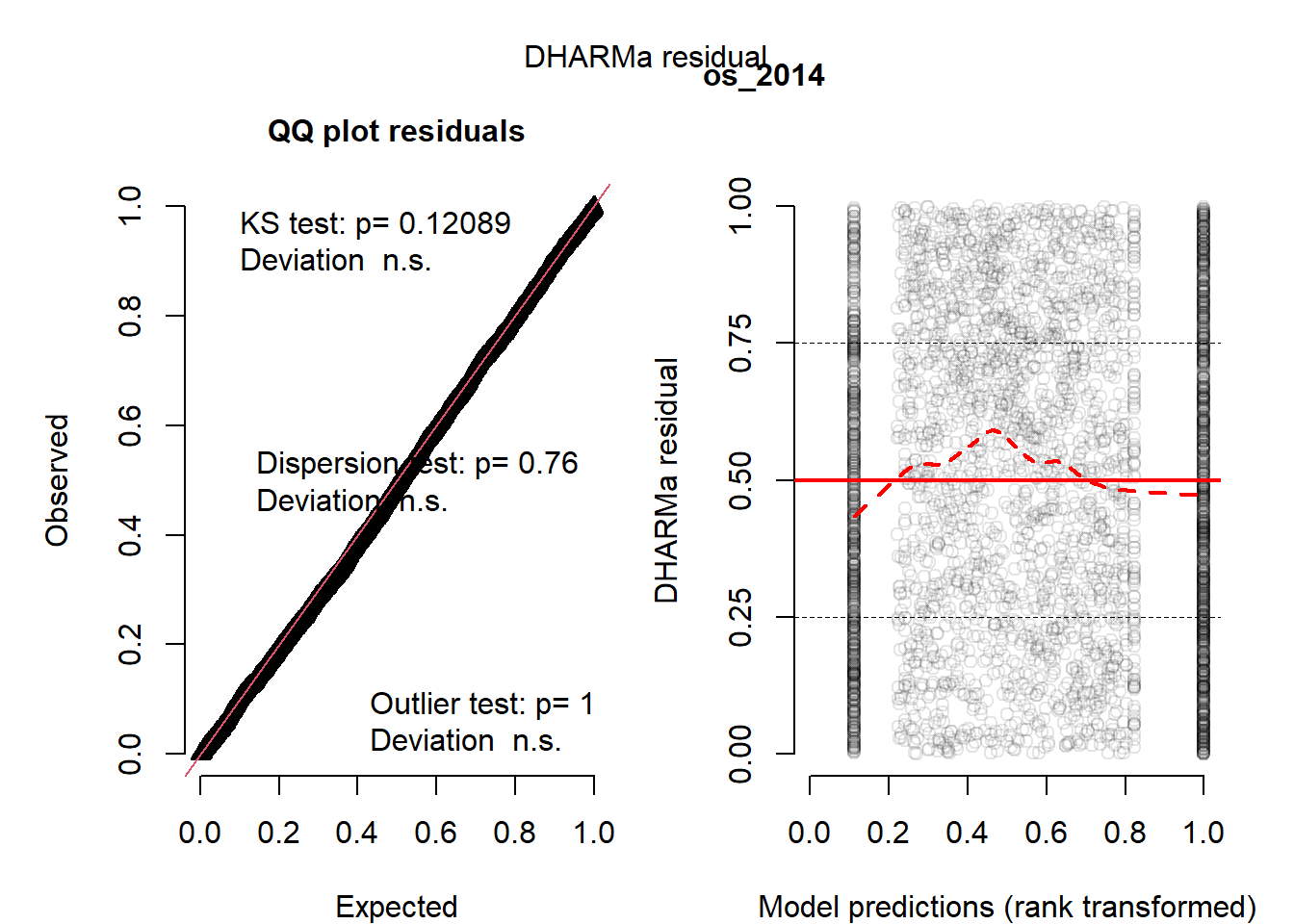
m <- glm(as.formula(paste0("pres_abs ~ ", v)), data = data, family = binomial)
# DHARMa Standardseite (QQ + Residuen vs Fitted)
sim <- simulateResiduals(m, n = 1000, plot = FALSE)
plot(sim, main = "") # Standardtitel ausblenden
mtext(v, side = 3, line = 0.5, font = 2) # eigene Überschrift (nur Variablenname)
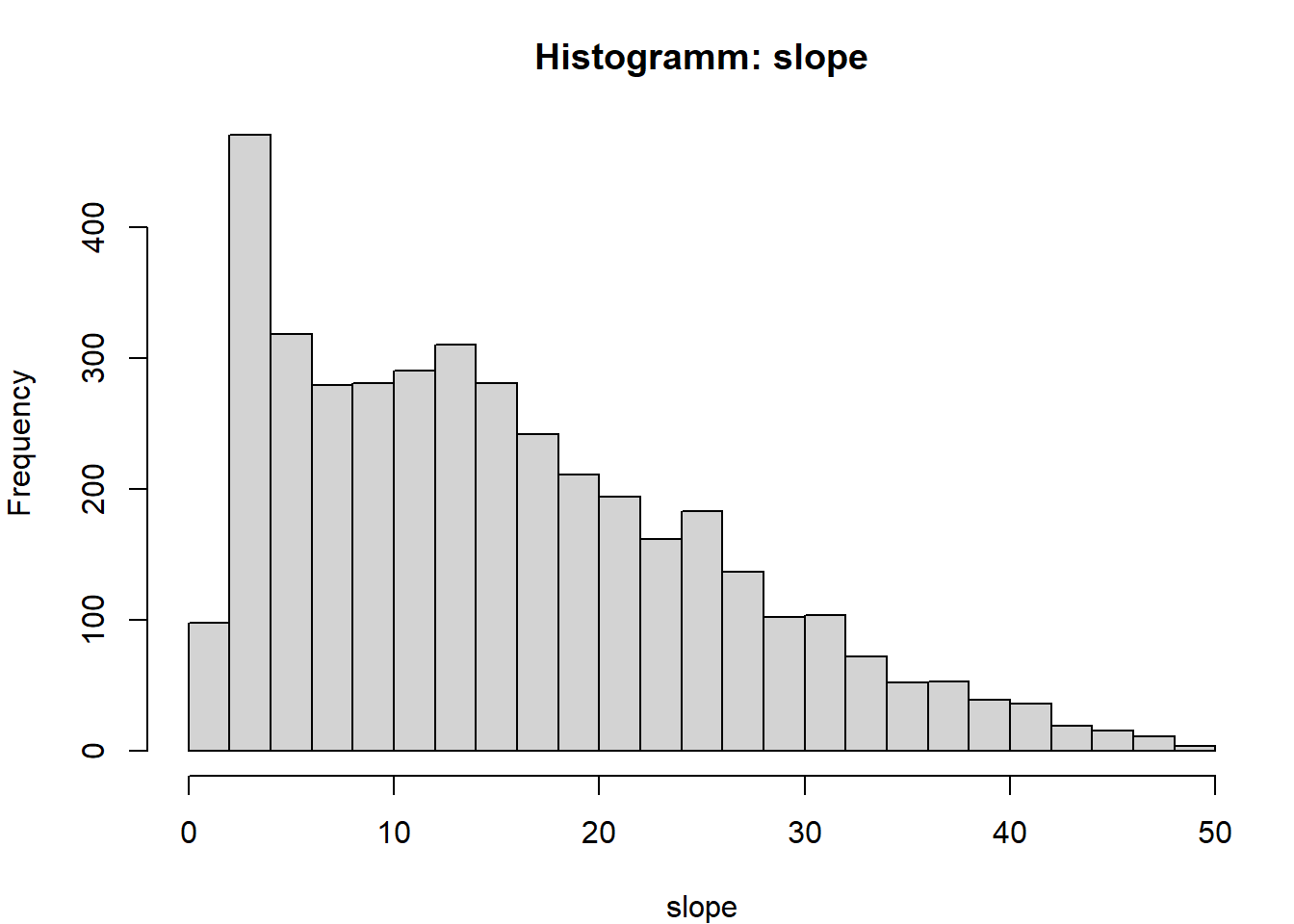
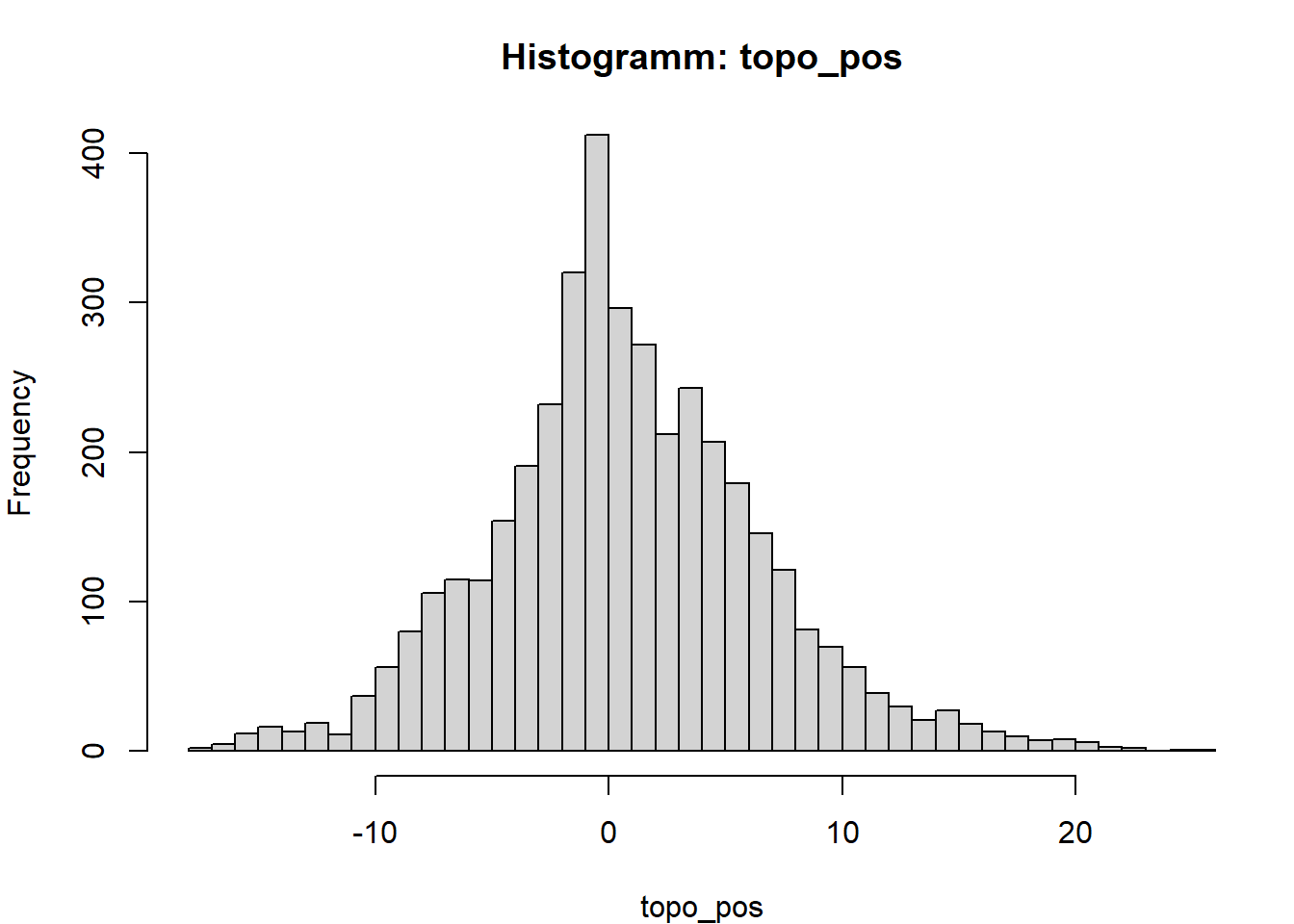
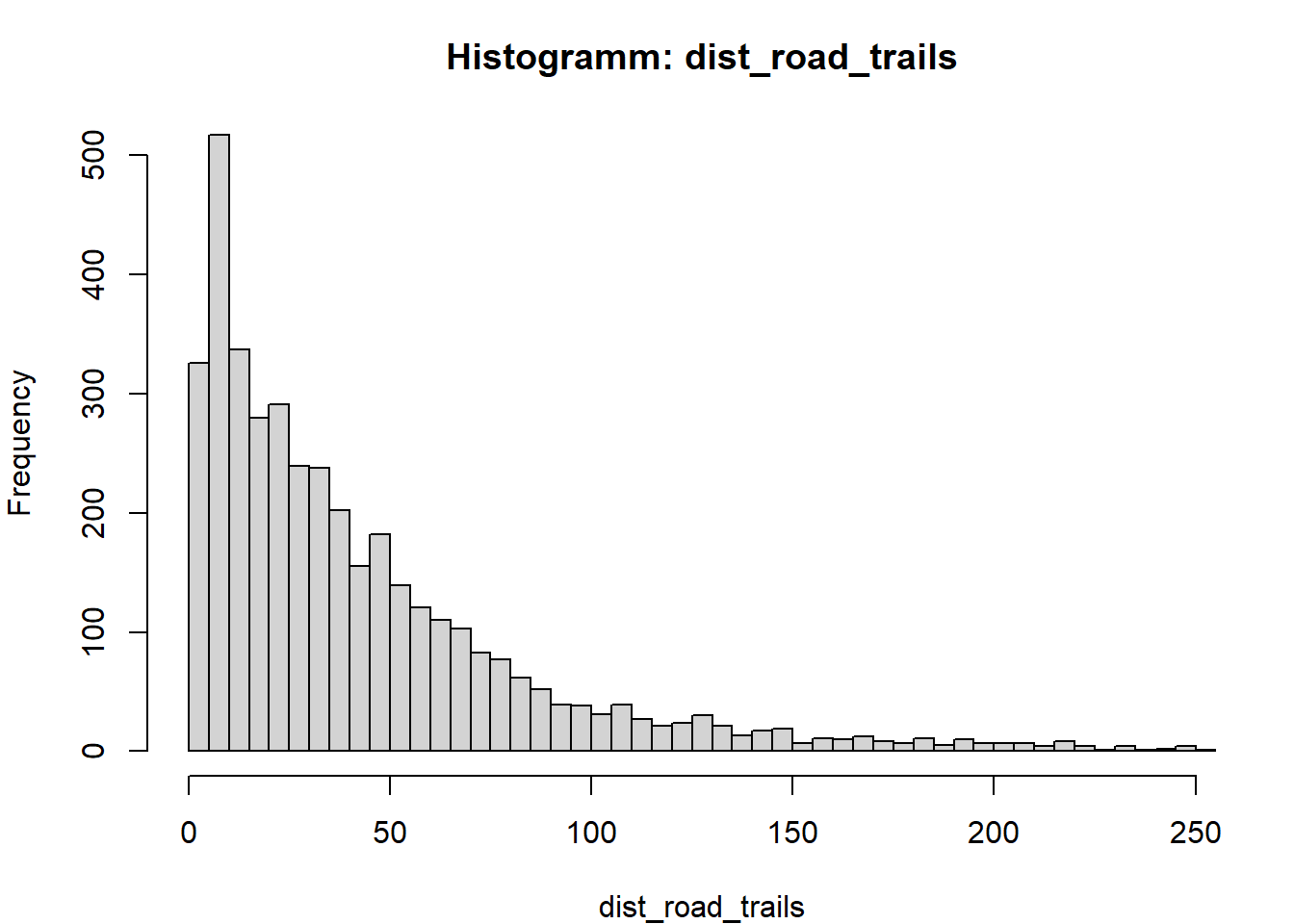
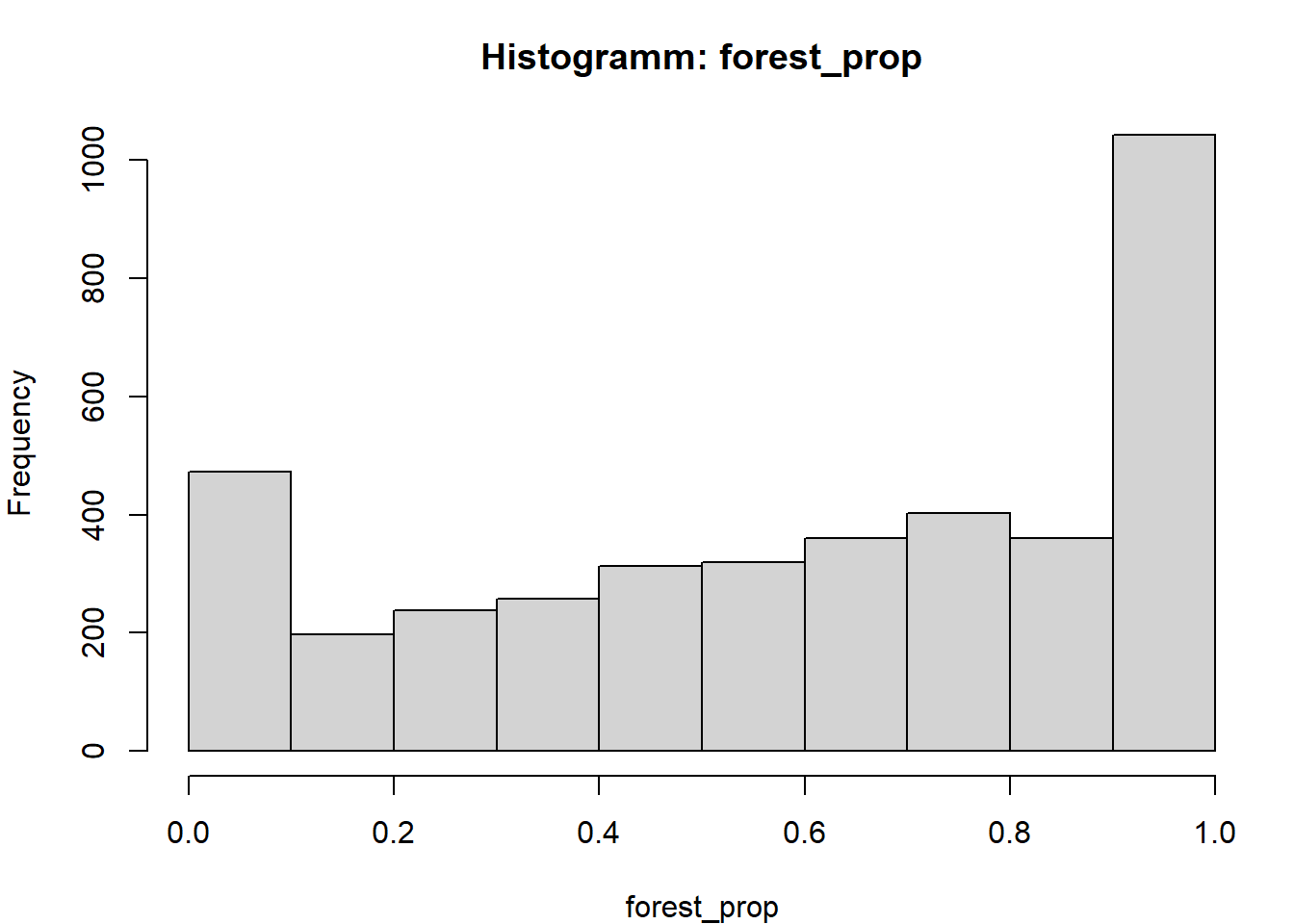
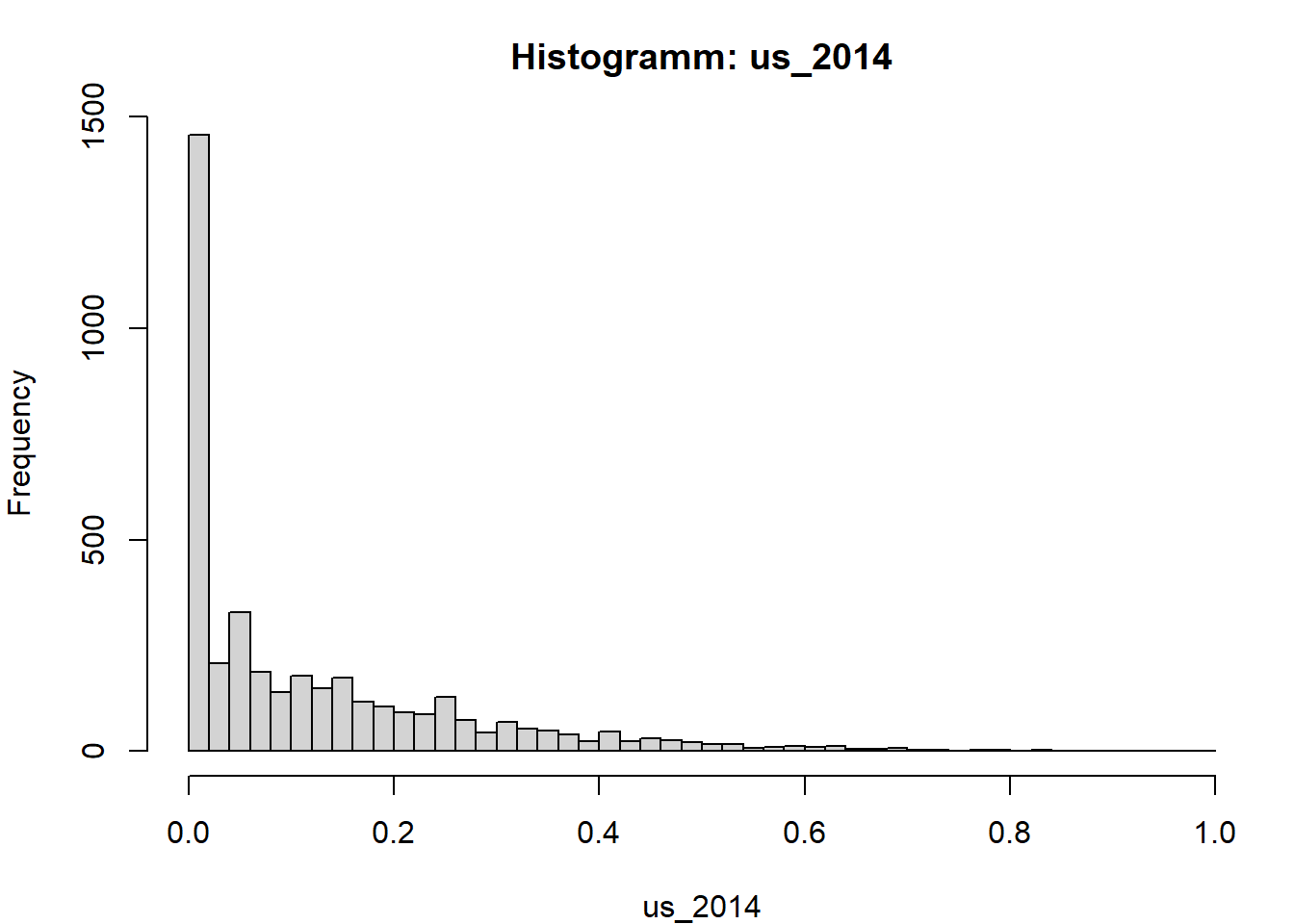
# Histogramm der Variable
par(mfrow = c(1,1), mar = c(4,4,3,1))
hist(x, breaks = "FD", main = paste("Histogramm:", v), xlab = v)
}
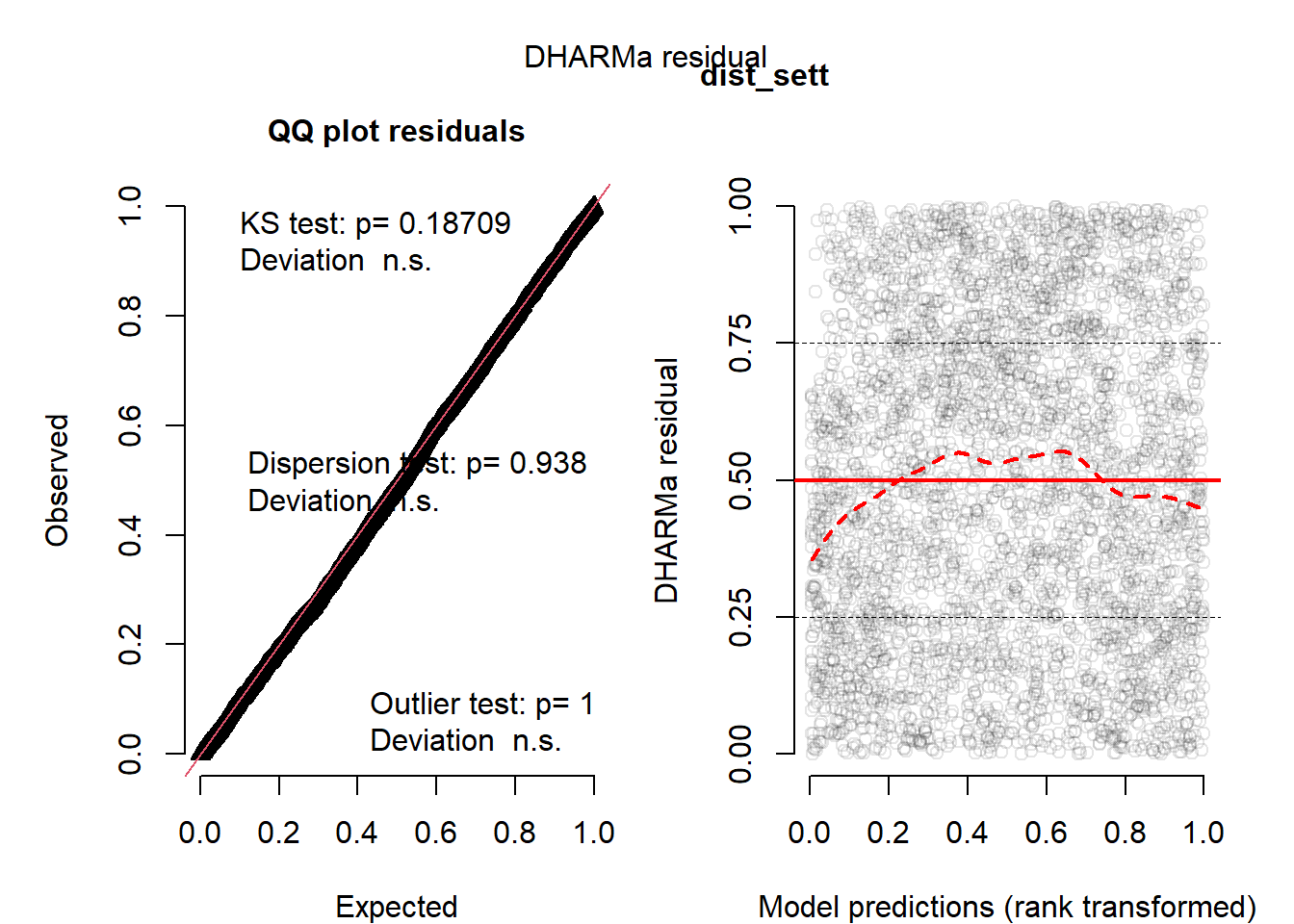
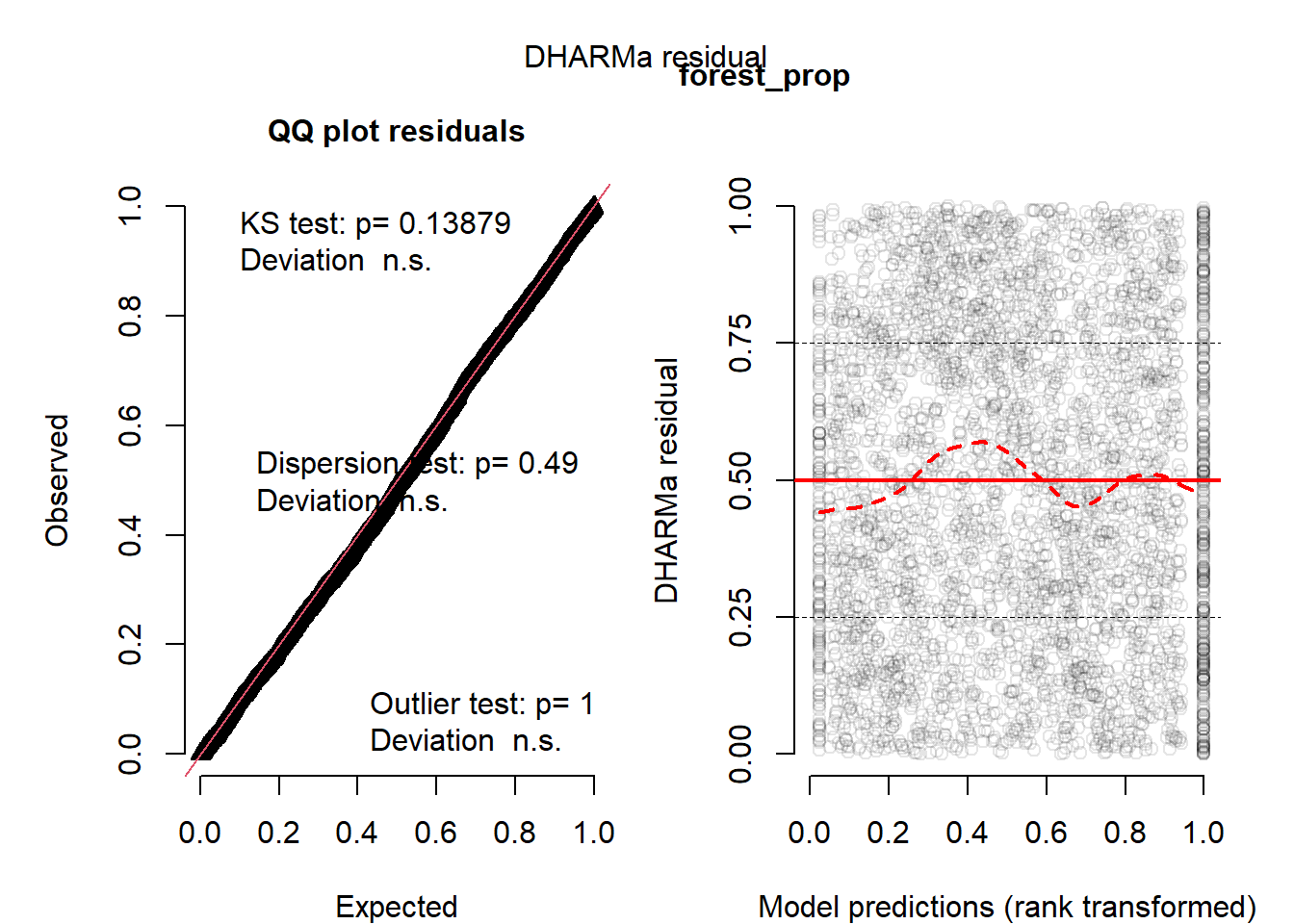
Aufgabe 5
Explorative Analysen der Variablen mit Scatterplots / Scatterplotmatrizen
- Zu Scatterplots und Scatterplotmatrizen gibt es viele verschiedene Funktionen / Packages, schaut im Internet und sucht euch eines welches euch passt.
- Testen der Korrelation zwischen den Variablen (Parametrisch oder nicht-parametrische Methode? Ausserdem: gewisse Scatterplotmatrizen zeigen euch die Koeffizenten direkt an)
Musterlösung
pairs.panels(DF_reh_day[5:12],
method = "pearson", # correlation method
hist.col = "#00AFBB",
density = TRUE, # show density plots
ellipses = TRUE # show correlation ellipses
)
Musterlösung
# Die Korrelation bei einem Teildatensatz zu testen reicht,
# denn die verwendeten Kreise sind die selben am Tag und in der Nacht,
# nur die Nutzung durch das Reh nicht.