library("sf")
library("dplyr")
library("ggplot2")
rotmilan <- read_sf("datasets/rauman/rotmilan.gpkg")
schweiz <- read_sf("datasets/rauman/schweiz.gpkg")
luftqualitaet <- read_sf("datasets/rauman/luftqualitaet.gpkg")Rauman 3: Übung C (Optional)
In dieser optionalen Übung wollen wir die G-Function für Luftqualitäts-Messstellen und Rotmilan Bewegungen berechnen und vergleichen.
Aufgabe 1
ggplot(rotmilan) +
geom_sf(data = schweiz) +
geom_sf(aes(colour = timestamp), alpha = 0.2) +
scale_color_datetime(low = "blue", high = "red")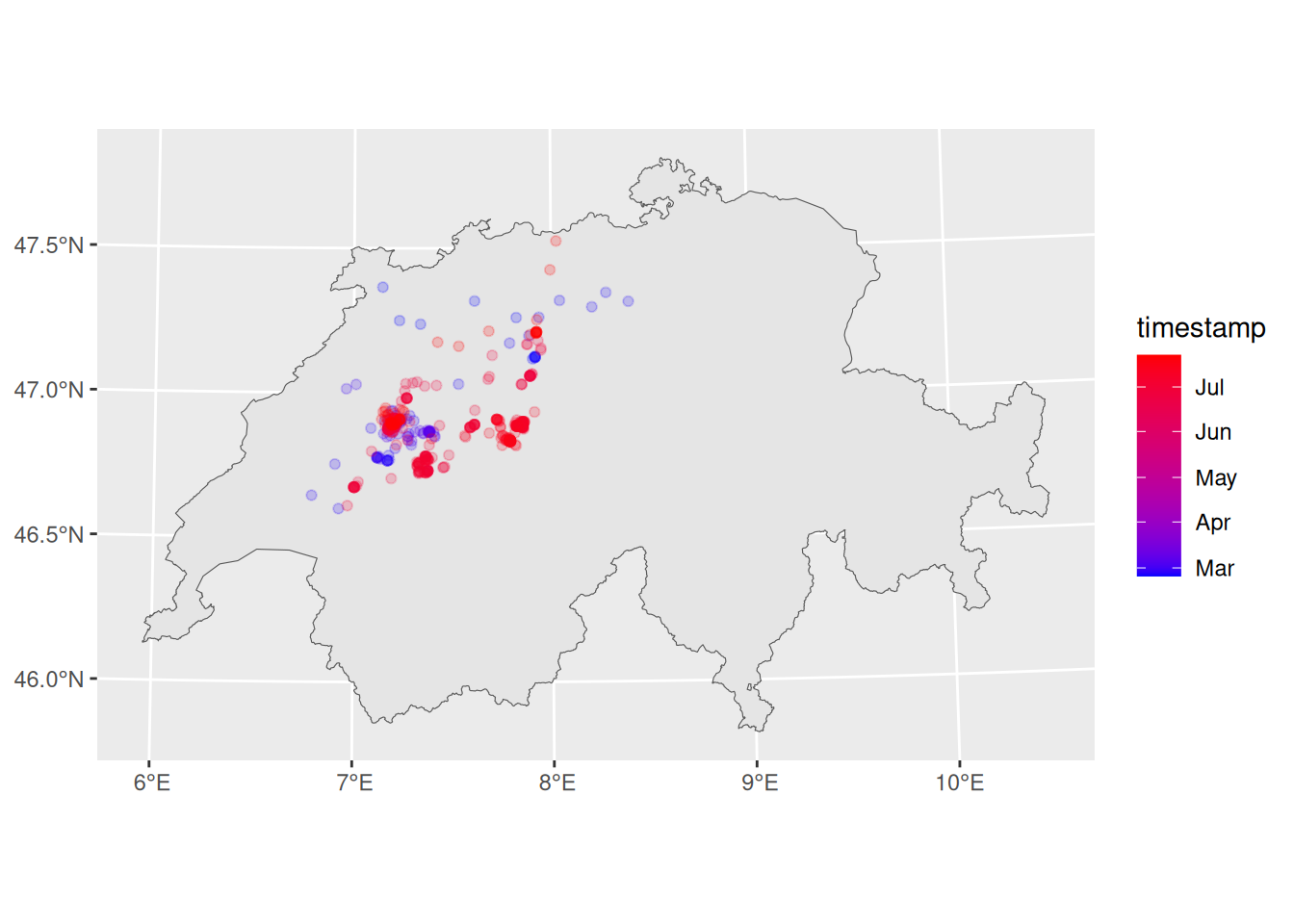
Aufgabe 2
Als erstes berechnen wir die G-Function für die Rotmilanpositionen:
Schritt 1
Mit st_distance() können Distanzen zwischen zwei sf-Datensätze berechnet werden. Wird nur ein Datensatz angegeben, wird eine Kreuzmatrix erstellt, wo die Distanzen zwischen allen Features zu allen anderen Features dargestellt werden. Wir nützen diese Funktion zur Berechnung der nächsten Nachbarn.
Musterlösung
rotmilan_distanzmatrix <- st_distance(rotmilan)
nrow(rotmilan_distanzmatrix)
ncol(rotmilan_distanzmatrix)
# zeige die ersten 6 Zeilen und Spalten der Matrix
# jeder Wert ist 2x vorhanden (vergleiche Wert [2,1] mit [1,2])
# die Diagonale ist die Distanz zu sich selber (gleich 0)
rotmilan_distanzmatrix[1:6, 1:6]Schritt 2
Nun wollen wir wissen, wie gross die kürzeste Distanz von jedem Punkt zu seinem nächsten Nachbarn ist, also die kürzeste Distanz pro Zeile. Bevor wir diese ermitteln, müssen wir die diagonalen Werte noch entfernen, denn diese stellen ja jeweils die Distanz zu sich selber dar und sind immer 0. Danach kann mit apply() eine Funktion (FUN = min) über die Zeilen (MARGIN = 1) einer Matrix (X = rotmilan_distanzmatrix) gerechnet werden. Zusätzlich müssen wir noch na.rm = TRUE setzen, damit NA Werte von der Berechnung ausgeschlossen werden. Das Resultat ist ein Vektor mit gleich vielen Werten wie Zeilen in der Matrix.
Musterlösung
diag(rotmilan_distanzmatrix) <- NA # entfernt alle diagonalen Werte
rotmilan_distanzmatrix[1:6, 1:6]
rotmilan_mindist <- apply(rotmilan_distanzmatrix, 1, min, na.rm = TRUE)Schritt 3
Nun müssen wir die Distanzen nach ihrer Grösse sortieren.
Musterlösung
rotmilan_mindist <- sort(rotmilan_mindist)Schritt 4
Jetzt berechnen wir die kummulierte Häufigkeit von jeder Distanz. Die kummulierte Häufikgeit vom ersten Wert ist 1 (der Index des ersten Wertes) dividiert durch die Anzahl Werte insgesamt. Mit seq_along erhalten wir die Indizes aller Werte, mit length die Anzahl Werte insgesamt.
Musterlösung
kumm_haeufgikeit <- seq_along(rotmilan_mindist) / length(rotmilan_mindist)Schritt 5
Nun wollen wir die kumulierte Häufigkeit der Werte in einer Verteilungsfunktion (engl: Empirical Cumulative Distribution Function, ECDF) darstellen. Dafür müssen wir die beiden Vektoren zuerst noch in einen Dataframe packen, damit ggplot damit klar kommt.
Musterlösung
rotmilan_mindist_df <- data.frame(
distanzen = rotmilan_mindist,
kumm_haeufgikeit = kumm_haeufgikeit
)
p <- ggplot() +
geom_line(data = rotmilan_mindist_df, aes(distanzen, kumm_haeufgikeit)) +
labs(x = "Distanz (Meter)", y = "Häufigkeit (kummuliert)")
p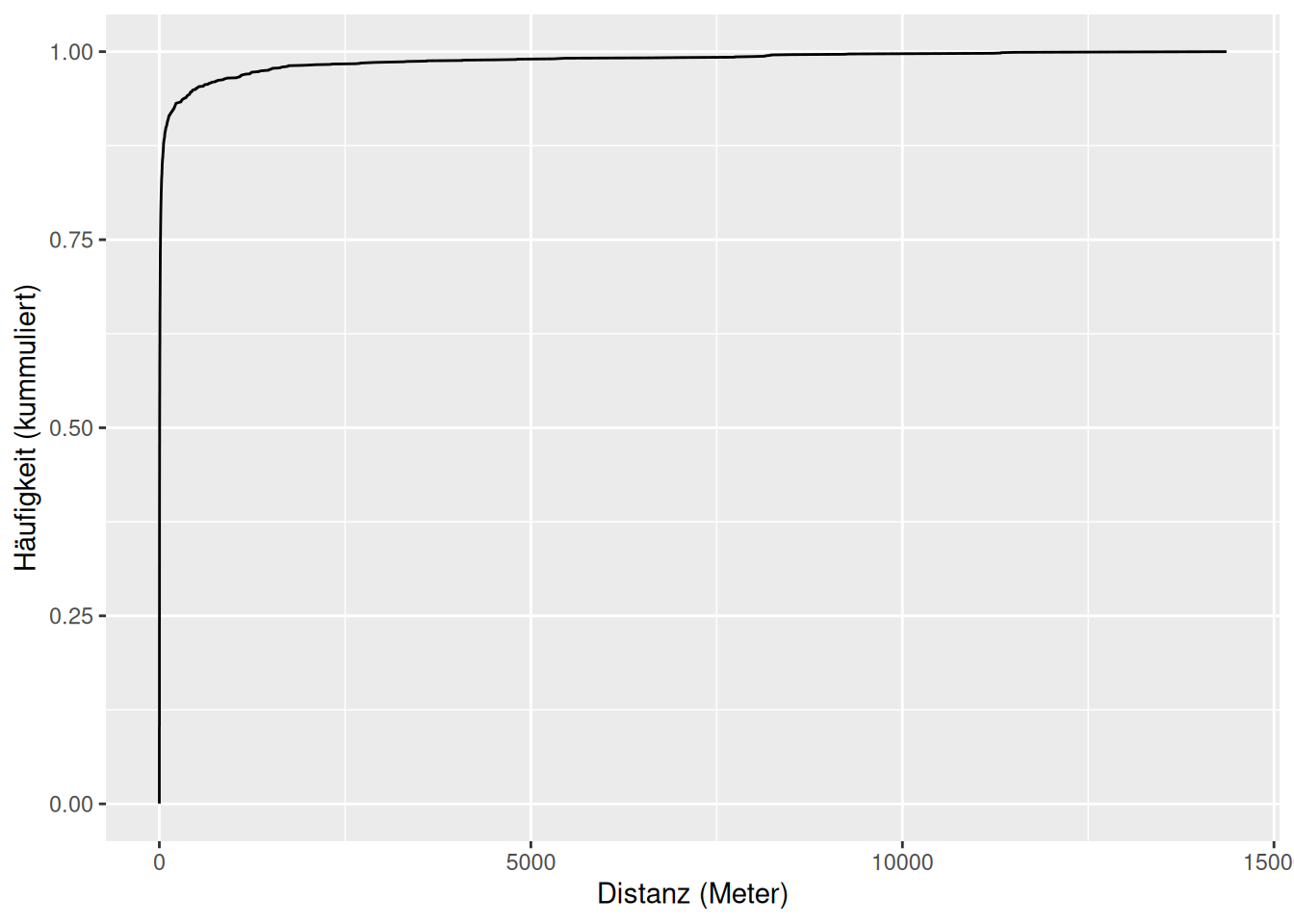
Lesehilfe:
Musterlösung
prob <- 0.95
res <- quantile(ecdf(rotmilan_mindist_df$distanzen), prob)
res2 <- quantile(ecdf(rotmilan_mindist_df$distanzen), 0.99)
xlim <- c(5000, NA)
ylim <- c(.5, .75)
p +
geom_segment(aes(x = res, xend = res, y = -Inf, yend = prob), colour = "lightblue") +
geom_segment(aes(x = -Inf, xend = res, y = prob, yend = prob), colour = "lightblue") +
geom_point(aes(x = res, y = prob), size = 3, colour = "lightblue") +
ggrepel::geom_label_repel(aes(x = 0, y = prob, label = paste0(prob * 100, "% der Werte...")),
xlim = xlim, ylim = ylim, hjust = 0, min.segment.length = 0, fill = "lightblue"
) +
ggrepel::geom_label_repel(aes(x = res, y = 0, label = paste0("... sind kleiner als ", round(res, 0), "m")),
xlim = xlim, ylim = ylim, hjust = 0, vjust = 1, fill = "lightblue", min.segment.length = 0, inherit.aes = FALSE
) +
scale_y_continuous(breaks = c(0, .25, .5, .75, prob, 1))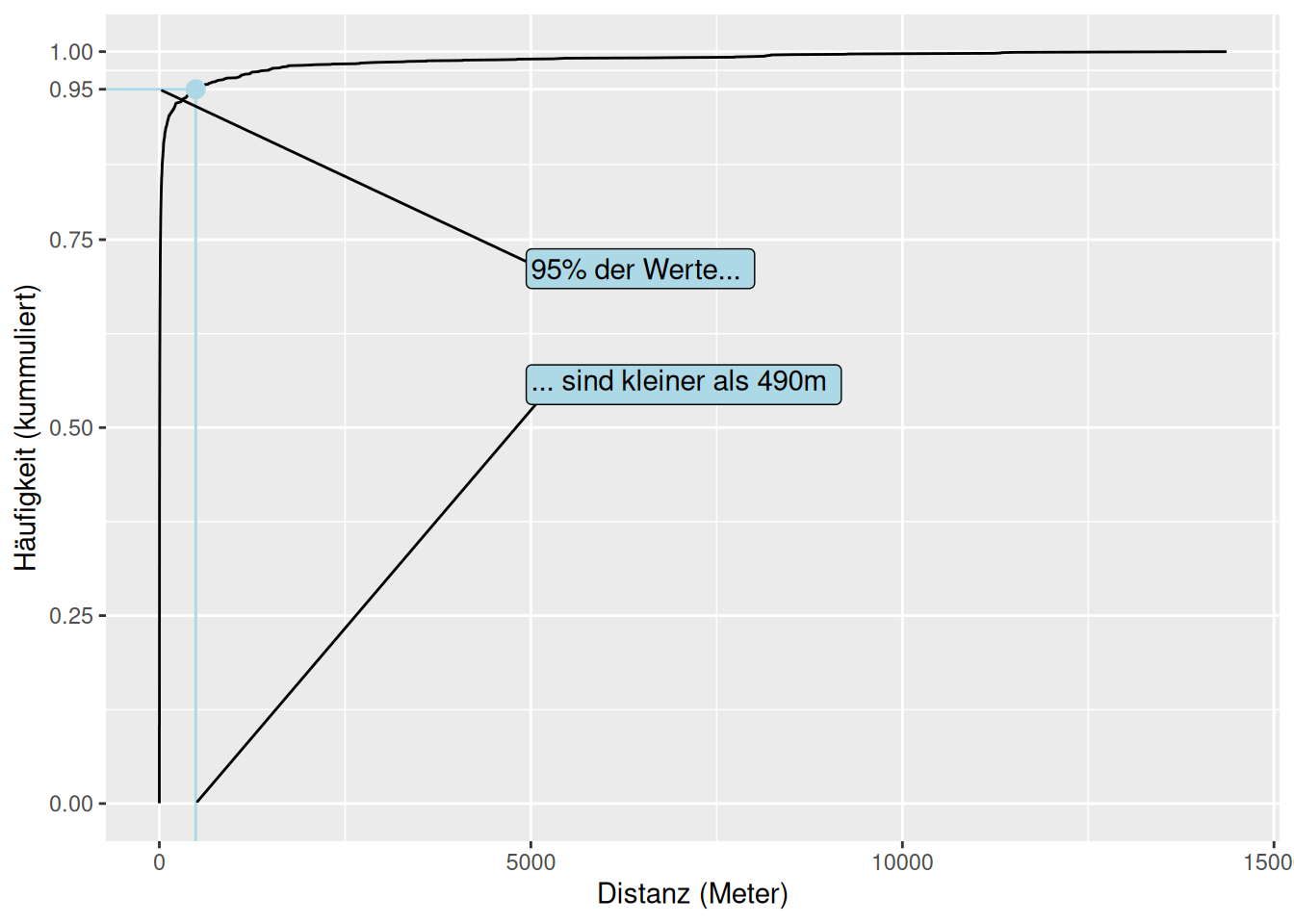
Aufgabe 3
Führe nun die gleichen Schritte mit luftqualitaet durch und vergleiche die ECDF-Plots.
Musterlösung
luftqualitaet_distanzmatrix <- st_distance(luftqualitaet)
diag(luftqualitaet_distanzmatrix) <- NA
luftqualitaet_mindist <- apply(luftqualitaet_distanzmatrix, 1, min, na.rm = TRUE)
luftqualitaet_mindist <- sort(luftqualitaet_mindist)
kumm_haeufgikeit_luftquali <- seq_along(luftqualitaet_mindist) / length(luftqualitaet_mindist)
luftqualitaet_mindist_df <- data.frame(
distanzen = luftqualitaet_mindist,
kumm_haeufgikeit = kumm_haeufgikeit_luftquali
)
luftqualitaet_mindist_df$data <- "Luftqualitaet"
rotmilan_mindist_df$data <- "Rotmilan"
mindist_df <- rbind(luftqualitaet_mindist_df, rotmilan_mindist_df)
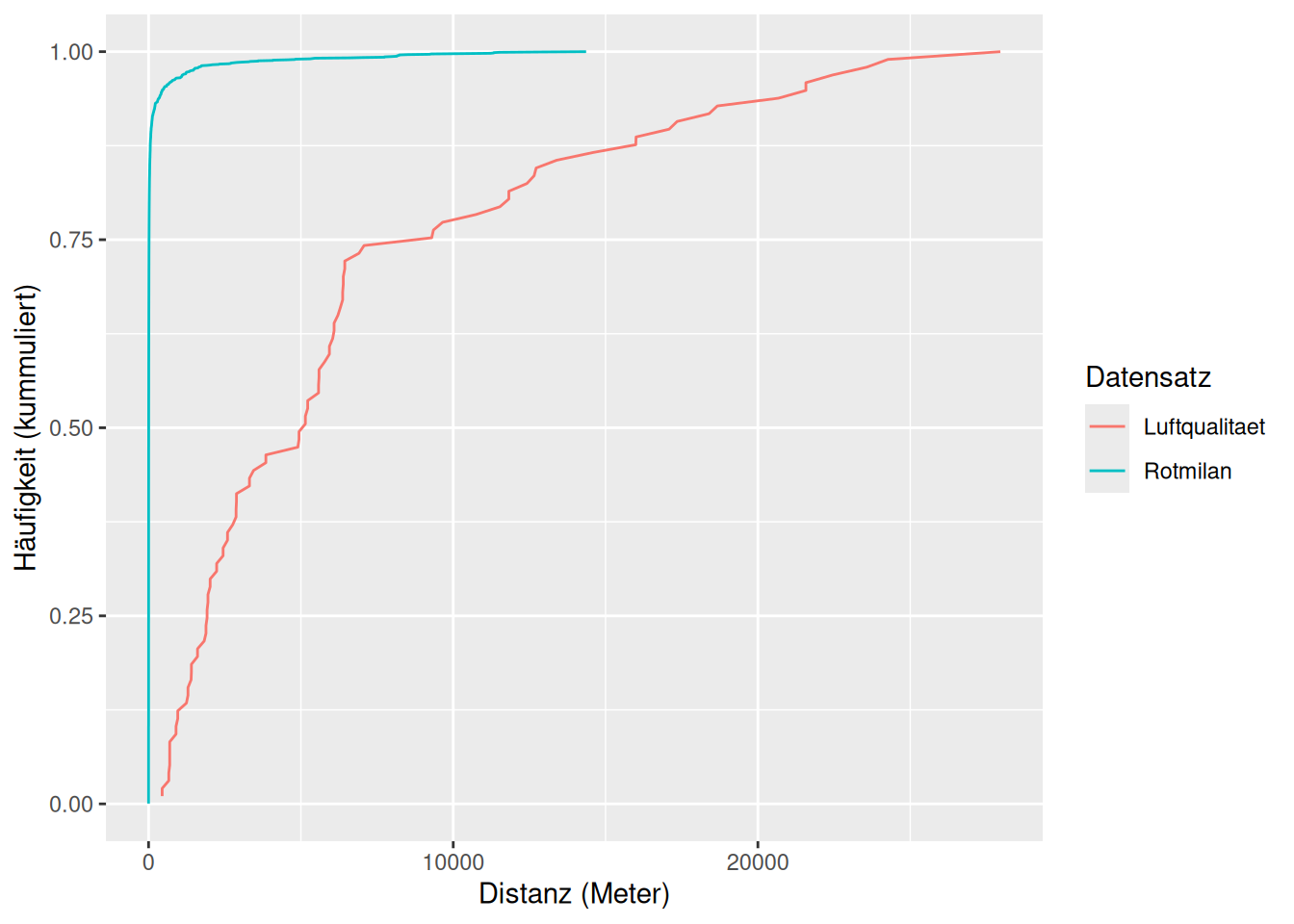
ggplot(mindist_df, ) +
geom_line(aes(distanzen, kumm_haeufgikeit, colour = data)) +
labs(x = "Distanz (Meter)", y = "Häufigkeit (kummuliert)", colour = "Datensatz")